巧用数据库 | 我是如何使用NCBI,UCSC,Ensembl,Uniprot四个数据库的?
我们吉凯基因网上商城(www.taogene.com)中引物产品对应的基因目前已经覆盖NCBI refseq数据库、mirbase数据库、circbase数据库中human,mouse,rat的所有基因以及Ensembl数据库的部分基因。
但今天呢,我们不说我们的引物产品,也不谈数据之间的差别,就说说这几个数据库到底能做什么?
毋庸置疑,NCBI,UCSC,Ensembl,UniProt四个数据库功能非常强大,下面给大家介绍下我自己用的最多的功能。
NCBI 中BLAST工具
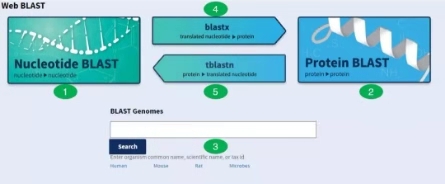
NCBI中的Nucleotide BLAST、Protein BLAST、BLAST Genomes(对应图中的1、2、3),这三种比对工具用的最多,其余两种blastx、tblastn(对应图中的4和5),用的相对较少,但是不得不说,真的好用!!
1.Nucleotide BLAST(BLASTN):nucleotide–nucleotide BLAST,核苷酸与核苷酸比对工具,可以序列之间比对,也可以与NCBI nucleotide database比对;
2.Protein BLAST(BLASTP):protein–protein BLAST,蛋白序列与蛋白序列比对工具。可以序列之间比对,也可以与NCBI Protein database比对;
3.BLAST Genomes:核苷酸与选择的基因组之间的比对;
4.blastx:核苷酸与蛋白序列比对,将给定的核酸序列按照六种阅读框架将其翻译成蛋白质与蛋白质数据库中的序列进行比对,对分析新序列和EST很有用;
5.tblastn:将给定的氨基酸序列与核酸数据库中的序列(双链)按不同的阅读框进行比对,对于寻找数据库中序列没有标注的新编码区很有用;
这五种比对工具相信大家都用过,那么比对结果怎么看呢?以tblastn举例。比对结果中需要注意Query Coverage和Identities两个数值,只有将两个数值结合起来看,才能很好地说明序列的比对情况。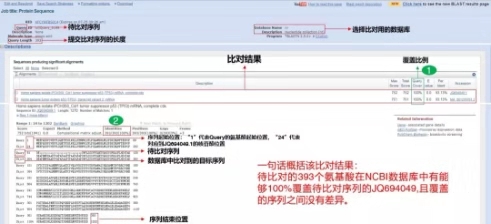

1.Query Coverage:对应图中的1,数值越高,代表与之匹配的序列越长;
2.Identities:对应图中的2,数值越大,代表与之同源性越高。
再举例一个概念:low-complexity sequence。顾名思义,就是序列复杂度低,比如连续的T,或者相对有规律的序列。这种序列在设计引物(比如qPCR引物)的时候尽量避开,原因是引物如果落在这些位置,容易导致引物的非特异性结合,带来的可能结果就是非特异性产物的产生。
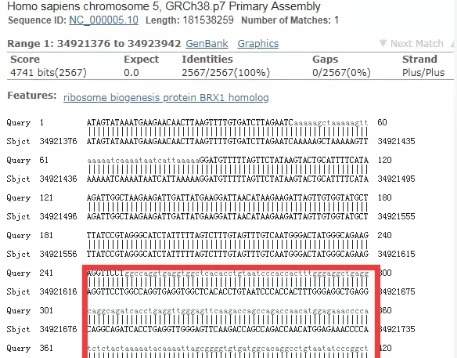
那如何从比对结果中确定哪部分序列是low-complexity sequence呢?如下图,比对结果中小写的、灰色的碱基序列就是low-complexity sequence。
UCSC
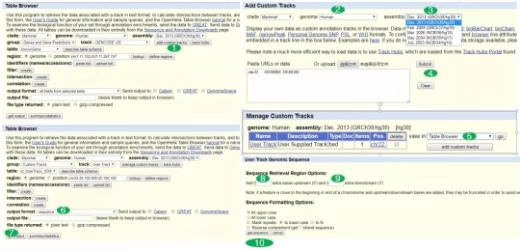
2. Table Browser可以很方便地根据基因的起始位置和终止位置获得对应的序列,同样可以选择不同的assembly。
Ensembl
TargetScan是最常用的microRNA与靶基因结合位点预测网站,该网站用的靶基因UTR数据库即Ensembl。
如下图,预测结果中给出了ENSMUST00000103114.2。首先从这个transcript可以看出,基因物种为mouse,带有version信息“.2”。由于Ensembl数据库信息是定期更新的,会有不同的release。即有可能ENSMUST00000103114.2 version的信息在数据库当前的release(Current Ensembl release 97)中并不存在,ENSMUST00000103114目前在Ensembl release 97中是ENSMUST00000103114.7 version。
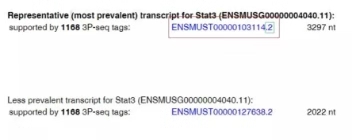
Current Ensembl release 97中ENSMUST00000103114的信息如下:
不同的release里边的序列可能会有不同,那如何找到ENSMUST00000103114.2对应的序列呢?进入到不同的release里边去找ENSMUST00000103114.2。
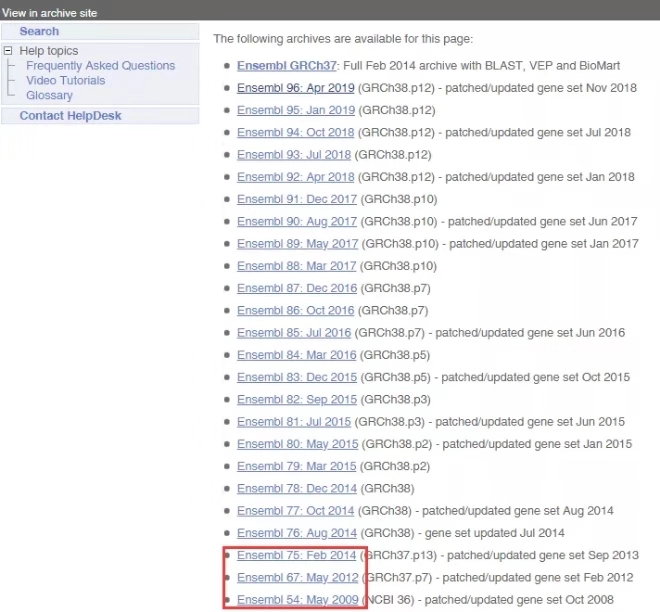
心明眼亮的你们可能注意到了一个问题,为什么没有release 66?Ensembl并不会把所有的release展示出来,那如果现有的release里边都没有ENSMUST00000103114.2,要去哪里找呢?去FTP下载(ftp://ftp.ensembl.org/pub/)即可。
UniProt
UniProt(Universal Protein Resource)是全球有关蛋白质方面信息最全面的资源库。UniProt提供了完全分类的、有丰富且准确注释信息的基于知识的蛋白质序列信息,数据库可以提供的信息包括蛋白功能描述、GO条目、细胞定位、组织特异性表达情况、生理病理情况描述、互作蛋白、Domain、翻译后修饰位点等信息。蛋白的信息描述段落均会标出引用文章,并且可以跳转到PubMed界面进行浏览。
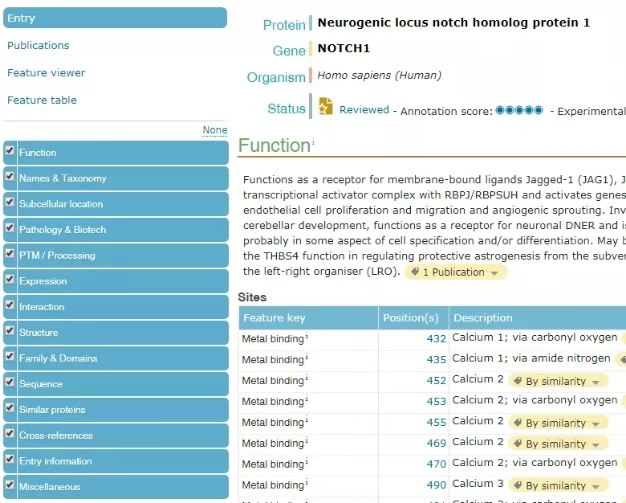
1.Function 板块可以看到基因的功能以及参与的生物学过程;
2.Names & Taxonomy板块可以看到基因的细胞定位以及拓扑结构域,比如NOTCH1在该板块能够看到胞外段,胞内段信息;
3.PTM / Processing板块描述了蛋白的翻译后修饰情况;
4.Structure板块可以看到蛋白的三维空间结构;
5.Sequence板块可以得到一个'canonical'序列信息,在不知道选择哪个转录本做研究室可以参考该信息判断。
