
Affymetrix SNP 芯片服务
产品名称: Affymetrix SNP 芯片服务
英文名称: Service of affymetrix SNP Array
产品编号:
产品价格: 询价021-51320288
产品产地: null
品牌商标: null
更新时间: 2024-12-19T16:11:10
使用范围: null
- 联系人 : 付先生
- 地址 : 浦东新区张江高科技园区李冰路151号
- 邮编 : 201203
- 所在区域 : 上海
- 电话 : 177****9967 点击查看
- 传真 : 点击查看
- 邮箱 : market@shbio.com
- 二维码 : 点击查看
Affymetrix SNP芯片服务
2005年4月29日,Affymetrix Inc.总裁苏珊西格尔女士率领代表团出席Affymetrix生物芯片上海服务中心的开张和揭牌仪式,标志着SBC成为国内第一个通过Affymetrix认证的,有资质的技术服务商。SBC为客户提供稳定而优质的Affymetrix SNP分析检测服务,为客户提供芯片杂交、洗涤、扫描和数据分析等服务。同时,我们还利用SBC与南方基因中心的合资子公司南方基因科技有限公司引进的Illumina微珠基因分型分析系统为广大科研工作者提供SNP芯片技术服务。 
近年来,SBC利用平台优势,为广大客户完成了多个项目,其中包括运用Affymetrix SNP 6.0芯片完成对狼人综合症疾病的遗传基础研究、Illumina芯片对银屑病易感基因的筛选等(详情请见参考文献)。
芯片推荐:Affymetrix Genome-Wide Human SNP Array 6.0
芯片介绍:
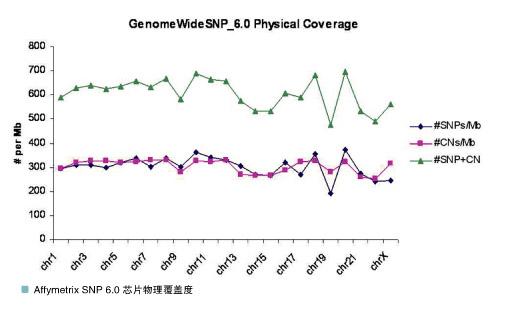
芯片介绍:这款芯片与Human SNP 6.0 芯片类似,既包含SNP探针,也包含CNV探针。一共涵盖623,124个SNP位点和916,269个CNV探针。
芯片介绍:芯片覆盖indica, aus, tropical japonica, temperate japonica, and group V (“aromatic”)多个水稻亚种的44,1000个SNP。
芯片特点:
1.全基因组覆盖:每10kb至少包括1个SNP,每个SNP设计12个探针技术重复相关系数R2 > 99%。
2.简便的样本:叶组织或抽提的DNA便可快速灵活的对遗传种质进行鉴定和分类。
3.从实验样本DNA到数据快速转换。
2. 样品浓度:浓度不低于55ng/µl。
3. 样品总量:每个样品总量不少于1µg。
4.样品溶剂:溶解在Reduced TE (10mM Tris, pH 8,0,0.1mM EDTA)中。
5. 样品运输: DNA低温运输(-20℃);在运输过程中请用parafilm将管口密封好,以防出现污染。
服务内容
1. DNA抽提;
2. 样品质检:用琼脂糖凝胶电泳或LAB-ON-CHIP系统对样品DNA进行质量检测和定量;
3. 实验过程:
a、扩增,标记; b、杂交、洗脱(具体见左图);
4. 图像扫描:用Affymetrix Scanner3000 7G激光共聚焦扫描仪对杂交结束后的芯片进行扫描;
5. 数据处理:确定检出的SNP位点相应的核苷酸。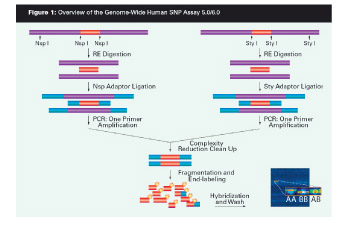
1. SNP位点过滤
1)Nocall rate>=10%
2)最小等位基因频率,Minor allele frequency<0.05
3)不符合哈维平衡的位点,HWE P value<0.001 的位点
1)样品群的分层分析
2)卡方检验、Fisher精确检验,比较各allele的频率分布在case和control两组中是否具有统计学差异
3)卡方检验、Fisher精确检验和Cochran-Armitage趋势检验,比较各基因型频率分布在两组中是否具有统计学差异
4. 单倍型分析
1)选取包含显著的SNP的区段进行单倍型分析,以进一步定位候选区段
2)频率较高的几种单倍型各自的频率,在case/control两组中的分布状况,卡方统计量及p值,判断单倍型与疾病相关性
结果报告
1、实验数据:原始数据(cel文件),基因芯片数据总表,SNP位点信息。
2、实验文件:SNP芯片实验操作方法。
3、实验图像:SNP芯片扫描图(jgp格式),所有样品的质检电泳图。
4、质检文件:DNA质检报告,SNP Call rate。
全基因组关联研究(Genome-wide association study,GWAS)是用来检测全基因组范围的遗传变异与可观测的性状之间的遗传关联的一种策略。在GWAS研究中的Manhattan图可以用来观测各个染色体中显著差异SNP探针的数据分布。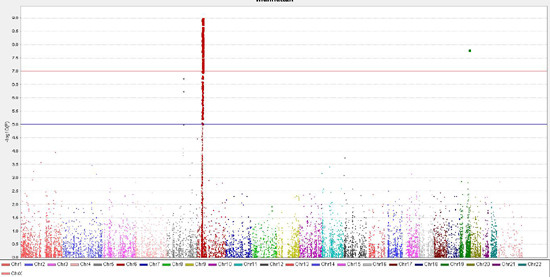
Ikram MK, Xueling S, Jensen RA, Cotch MF, Hewitt AW, et al. (2010) Four Novel Loci (19q13, 6q24, 12q24, and 5q14) Influence the Microcirculation In Vivo. PLoS Genet 6(10): e1001184. doi:10.1371/journal.pgen.1001184.
每条染色体上minP的分布,横坐标为染色体上位置, 纵坐标为-log10(minP), 每个点代表一个通过筛选的SNP, 位于棕色线上方的点minP<0.001。以2号染色体为例。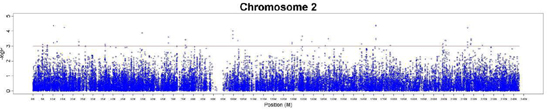
以2号染色体上的某一区段为例。
连锁不平衡(linkage disequilibrium)是指基因组中不同基因座间存在的非随机关联,即不同基因座的非等位基因间的非随机组合。LD Plot表示该基因所有snp的的连锁情况,各个方块的颜色由浅至深(白—红),表示连锁程度由低到高,深红色表示完全连锁。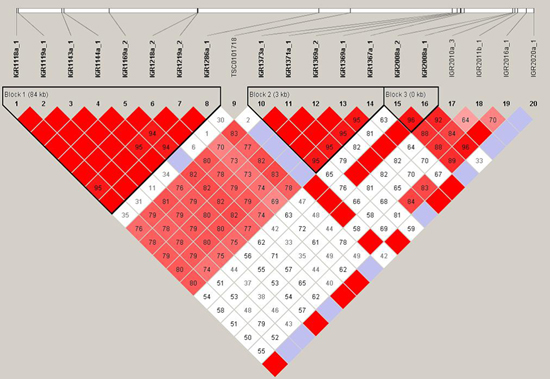
LOD plot
LOD(log odds score), 优势对数记分法.是根据遗传标志与致病基因的连锁,和在家系中的重组值,即两者之间的遗传距离,得出两者连锁的似然性比例。Lod值为0,意味着连锁假设与不连锁假设的可能性相等;Lod值为正值,有利于连锁;Lod值为负值,表示有一定重组率的连锁。显著的域值是﹢3和﹢2。Lod﹦﹢3时,连锁的概率为95%。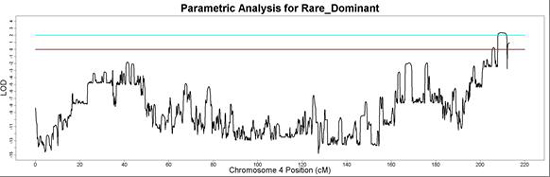
单倍型又称单体型,是tagSNP的call在染色单体上的线性排列,单倍型分析的目的是看是否所有患病个体都继承了同样的单体型。在下图中,患病个体画框的里面单体型是一样的。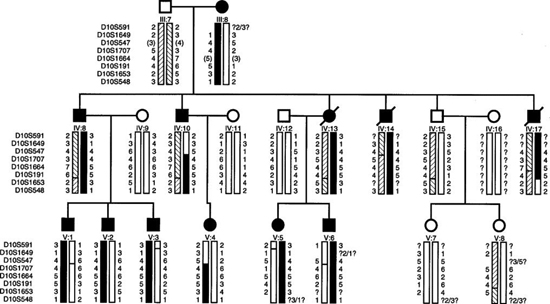
2. HUGO Pan-Asian SNP Consortium, Abdulla MA, Ahmed I, Assawamakin A, Bhak J, Brahmachari SK, Calacal GC, Chaurasia A, Chen CH, Chen J, Chen YT, Chu J, Cutiongco-de la Paz EM, De Ungria MC, Delfin FC, Edo J, Fuchareon S, Ghang H, Gojobori T, Han J, Ho SF, Hoh BP, Huang W, Inoko H, Jha P, Jinam TA, Jin L, Jung J, Kangwanpong D, Kampuansai J, Kennedy GC, Khurana P, Kim HL, Kim K, Kim S, Kim WY, Kimm K, Kimura R, Koike T, Kulawonganunchai S, Kumar V, Lai PS, Lee JY, Lee S, Liu ET, Majumder PP, Mandapati KK, Marzuki S, Mitchell W, Mukerji M, Naritomi K, Ngamphiw C, Niikawa N, Nishida N, Oh B, Oh S, Ohashi J, Oka A, Ong R, Padilla CD, Palittapongarnpim P, Perdigon HB, Phipps ME, Png E, Sakaki Y, Salvador JM, Sandraling Y, Scaria V, Seielstad M, Sidek MR, Sinha A, Srikummool M, Sudoyo H, Sugano S, Suryadi H, Suzuki Y, Tabbada KA, Tan A, Tokunaga K, Tongsima S, Villamor LP, Wang E, Wang Y, Wang H, Wu JY, Xiao H, Xu S, Yang JO, Shugart YY, Yoo HS, Yuan W, Zhao G, Zilfalil BA; Indian Genome Variation Consortium. Mapping Human Genetic Diversity in Asia. Science. 2009, VOL 326: 1541-1545.
3.Jian-Wen Han,Hou-Feng Zheng,Yong Cui,Liang-Dan Sun,Dong-Qing Ye,Zhi Hu,Jin-Hua Xu,Zhi-Ming Cai,Wei Huang,Guo-Ping Zhao,Hong-Fu Xie,Hong Fang,Qian-Jin Lu,Jian-Hua Xu,Xiang-Pei Li,Yun-Feng Pan,Dan-Qi Deng,Fan-Qin Zeng,Zhi-Zhong Ye,Xiao-Yan Zhang,Qing-Wen Wang,Fei Hao,Li Ma1,Xian-Bo Zuo,Fu-Sheng Zhou,Wen-Hui Du,Yi-Lin Cheng,Jian-Qiang Yang,Song-Ke Shen,Jian Li,Yu-Jun Sheng,Xiao-Xia Zuo,Wei-Fang Zhu,Fei Gao,Pei-Lian Zhang,Qing Guo,Bo Li,Min Gao,Feng-Li Xiao,Cheng Quan,Chi Zhang,Zheng Zhang,Kun-Ju Zhu,Yang Li,Da-Yan Hu,Wen-Sheng Lu,Jian-Lin Huang,Sheng-Xiu Liu,Hui Li,Yun-Qing Ren,Zai-Xing Wang,Chun-Jun Yang,Pei-Guang Wang,Wen-Ming Zhou,Yong-Mei Lv,An-Ping Zhang,Sheng-Quan Zhang,Da Lin,Yi Li,Hui Qi Low,Min Shen,Zhi-Fang Zhai1,Ying Wang,Feng-Yu Zhang,Sen Yang,Jian-Jun Liu& Xue-Jun Zhang.Genome-wide association study in a Chinese Han population identifies nine new susceptibility loci for systemic lupus erythematosus.Nature Genetics.18 October 2009|doi:10.1038/ng.472
