
空间转录组测序│空间基因表达
产品名称: 空间转录组测序│空间基因表达
英文名称: 10X Genomics Visium
产品编号: 10X Genomics Visium
产品价格: 电话咨询:021-58955370
产品产地: null
品牌商标: null
更新时间: 2024-01-26T09:44:58
使用范围: null
- 联系人 : 付先生
- 地址 : 浦东新区张江高科技园区李冰路151号
- 邮编 : 201203
- 所在区域 : 上海
- 电话 : 177****9967 点击查看
- 传真 : 点击查看
- 邮箱 : market@shbio.com
- 二维码 : 点击查看
空间转录组(Spatial Transcriptomics)就是将基因的表达情况与关注的组织切片的免疫化学染色图像进行整合,从而将组织内不同细胞的基因表达信息定位到组织的原始空间位置上去,进而直接观测组织中不同部位功能区基因表达的差异。空间转录组技术利用了常规的原位技术和组学技术两方面的优势。
10X Genomics Visium 技术优势
空间转录组学技术是转录组学研究领域的新方向,也是研究细胞异质性方面的新方法。
(1)准确定位:探针有效定位组织中 RNA 空间位置,实时了解组织中的转录组天然状态。
(2)高效易行:芯片设计简单易操作、重复性高、可快速获得高分辨率空间转录组信息。
(3)适用性广:有效应用于发育生物学、肿瘤生物学、脑神经科学、植物研究等各生物学领域。
10X Genomics Visium 技术原理
将冷冻组织切片放置在 10X Genomics Visium 芯片的捕获区域内,进行 HE 染色和成像后,对组织切片进行透化处理,细胞内的 mRNA 释放,从而被芯片上带有 oligo-dT 的探针捕获,并且每个探针都带有特异的位置信息(Spatial barcode),然后以 mRNA 为模版进行 cDNA 合成,构建文库后再通过测序,获得基因表达信息的同时,每一条测序 reads 因带有 Spatial barcode,从而能够获得基因表达的位置信息。
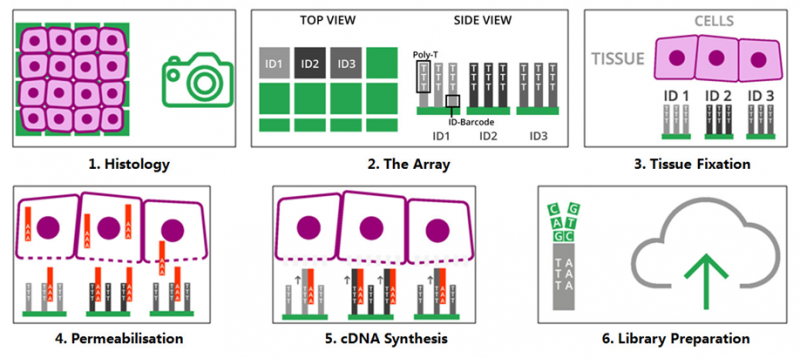
图 2 10X Genomics Visium 空间转录组技术的原理
10X Genomics Visium 实验流程
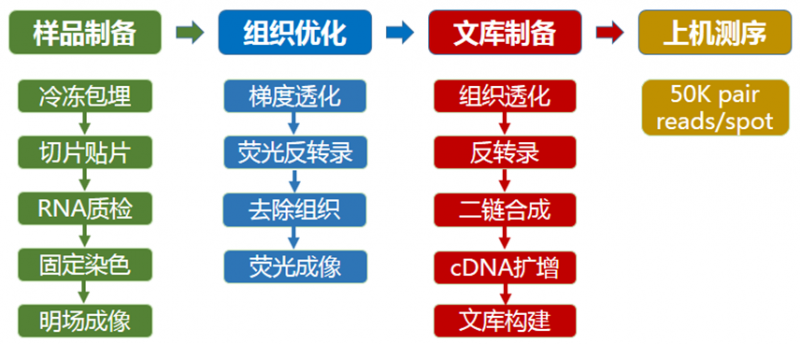
图 5 10X Genomics Visium 实验流程
第一步:样品制备
1、新鲜组织样本速冻和 OCT 包埋
成功制备样品是空间转录组实验成功的关键,要点是尽可能不让组织中 RNA 降解。首先,新鲜样本取材时就要记好其空间方向,而后使用 PBS 冲洗,除去残留血液,利用实验室用纸吸干组织表面多余的液体,防止冷冻后冰晶的形成。用预冷的镊子或刮刀将组织完全浸没于已在液氮中预冷 15min 的异戊烷里,直至完全冰冻(~1min)。包埋盒上需标记组织样本的方向,用冷却的 OCT 铺平包埋盒底部(放在干冰上使其冻结),将冰冻后的组织,放置在包埋盒中心位置,继续倒入 OCT 完全覆盖样本组织(组织周围不要产生气泡)。立即将含有组织和 OCT 的包埋盒放在干冰粉上,直至完全冻结(约 30min 以上)。将包埋盒放置到密封袋中,干冰运输。
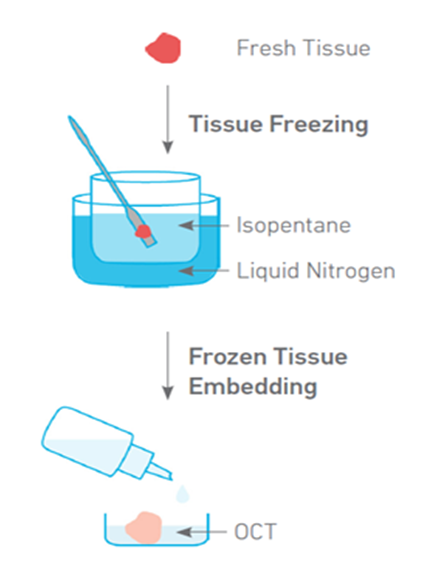
图 6 组织速冻和 OCT 包埋示意图

图 7 组织速冻和 OCT 包埋
2、冷冻切片切、组织样本质控及贴片
冷冻切片机腔体温度设定为–20℃,将冻头的温度设定为–10℃。在切片之前,必须将 OCT 包埋的组织块放在冷冻切片机腔体中温度平衡 30 分钟以上。在样本台上安装 OCT 包埋组织块,通过冷冻切片去除过量的 OCT 直到组织可见,切片时可以对较大的组织样本进行分割,切割后生成较小的样本以覆盖捕获区域。取 10 片组织切片进行 RNA 抽提并质检,确定组织中 RNA 完整性(RIN>7)。
对于大多数组织类型,建议的切片厚度为 10 µm,如果切片出现裂纹,则说明冻头太冷,如果切片出现褶皱,则说明冻头太热,相应地调节温度。获得所需的组织切片后,用冷冻切片机的刷子轻轻刷 OCT 的周围,将其小心地刷至平整。将切片放置在预先平衡温度的芯片上,立即将手指放在芯片的背面几秒钟,确保整个组织切片都完全粘附在芯片上,然后立即将其放在不锈钢冷冻室上以冻结切片。在切片和组织放置期间,请勿在任何时候从冷冻切片机中取出芯片。用 OCT 覆盖裸露的组织并使其冻结,保存在–80ºC 的密封容器中。

图 8 组织切片及贴片
第二步:组织优化
组织优化芯片包含 8 个捕获区域,其中 6 个区域分别设置 6 个不同的透化时间梯度,另外两个区域 1 个为不加透化剂的阴性对照,另 1 个为阳性对照,不放组织切片,而是直接加入 RNA。其实验流程为:固定→染色→明场拍照→组织透化→荧光 cDNA 合成→组织移除→荧光扫描,根据荧光强度判断最优(最亮)的透化时间。
1、甲醇固定
将芯片放在热循环仪适配器上,在 37°C 下孵育 1 分钟。然后将芯片完全浸入预冷的甲醇中在 -20°C 孵育 30 分钟。
2、H&E 染色
滴加 500μl 异丙醇,室温孵育 1 分钟;将芯片上试剂去除,风干芯片,加入 1 ml 苏木精,室温孵育 7 分钟。将芯片上试剂去除,然后将芯片浸入水中若干次,加入 1 ml Bluing Buffer,室温孵育 2 分钟。将芯片上试剂去除,将芯片浸入水中 5 次。加入 1 ml 伊红混合物,室温孵育 1 分钟。将芯片上试剂去除,将芯片浸入水中 15 次,风干直至组织不透明。在 37°C 下孵育芯片 5 分钟后,进行明场成像实验。

图 9 甲醇固定与 HE 染色
3、组织透化
将芯片放在芯片盒中,阳性和阴性对照均不加透化酶。在梯度时间最长的孔中添加 70µl 透化酶,放置在 37°C 的 PCR 适配器上。6 分钟后,向梯度时间次长孔中添加 70µl 通透酶,并在 37°C 的 PCR 适配器上孵育,重复此过程,直至加到最短的孵育时间。从每个孔中除去透化酶,阳性对照以外的所有孔中添加 100µl 0.1X SSC,从每个孔中移出 0.1X SSC。向每个孔中加入 50µl Fluorescent RT Master Mix,将其放在预热的 PCR 仪(设置条件)以启动 cDNA 合成。从孔中吸出 Fluorescent RT Master Mix,向每个孔中添加 100µl 0.1X SSC,从每个孔中吸出 0.1X SSC。向每个孔中加入 70µl Tissue Removal Mix,然后放在 PCR 仪适配器上孵育,从孔中吸出 Tissue Removal Mix。从芯片盒中取出芯片,浸入预热的 2X SSC-0.1%SDS 中 15 次,浸入 0.2X SSC 中 15 次,浸入 0.1X SSC 中 15 次。在载片旋转器中离心 30s,确认芯片上没有剩余的组织。
4、成像
应使用 Visium Imaging Test Slide 验证成像设置。明场成像基准框和基准标记应清晰可见,并使用 Brightfeld 设置聚焦。Visium Imaging Test Slide 四个区域(A1,B2,C1,D2)具有荧光斑点,可通过 TRITC 和 Cy5 flter cubes 检测到,荧光设置应清晰可见 A1,B2,C1 和 D2 中的荧光点,且荧光点信号应从左到右减小。
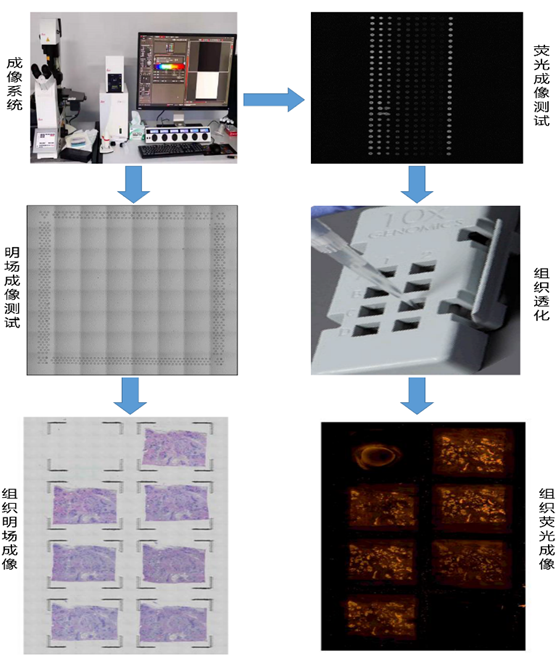
图 10 明场成像和荧光成像
第三步:正式实验
正式实验用的基因表达芯片上有 4 个捕获区域,其实验流程为:切片及贴片→甲醇固定→HE 染色→明场拍照→透化(最佳透化时间)→cDNA 合成→第二链 cDNA 合成→变性→循环数确定→cDNA 扩增→纯化和质控→片段化,末端修复,加 A→连接头→样本 index PCR→上机测序。前 5 步和组织优化过程大同小异,只是所用芯片不同,下图给出 cDNA 合成及扩增示意图。
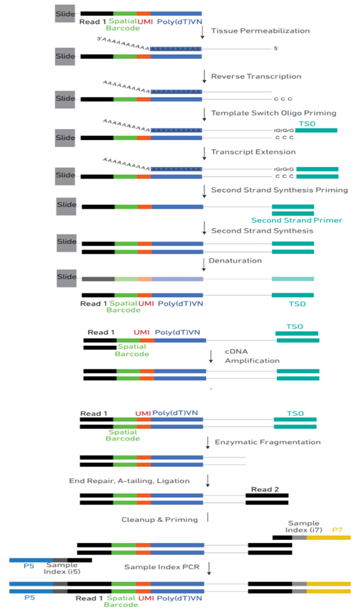
图 11 cDNA 合成、扩增及文库制备流程图
10X Genomics Visium 数据分析
空间转录组数据分析的核心是根据每个芯片上每个 spot 的基因表达信息进行聚类,然后将 spot 根据坐标位置序列放回到组织的图像上,同时可以对每个 gene 在组织上表达的空间位置进行定位。
获得测序数据后,首先利用 Space Ranger 软件可以自动化的对图像进行处理、数据比对和 Barcode 处理。另外一个软件 Loupe Cell Browser 是一个适用于 Windows 和 MacOS 的桌面应用程序,它可以快速、轻松地可视化和分析 10X Visium 数据。伯豪生物除了提供 spot 基因数和 UMI 数统计、切片 spot 聚类和聚类亚群 marker 基因分析等基础和高级分外,同时还提供个性化分析,如特定 pathway 功能富集分析等。
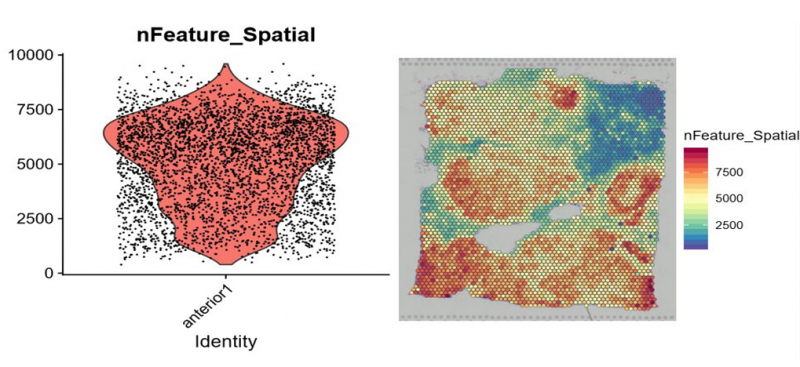
图 12 每个 spot 特异表达的基因数统计
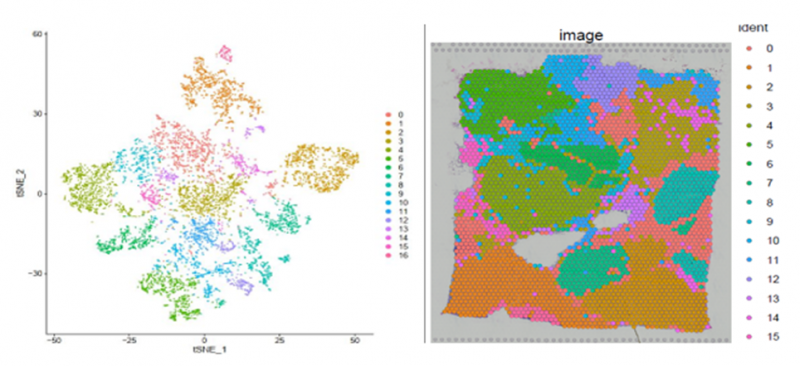
图 13 聚类结果及切片 spot 位置分布展示
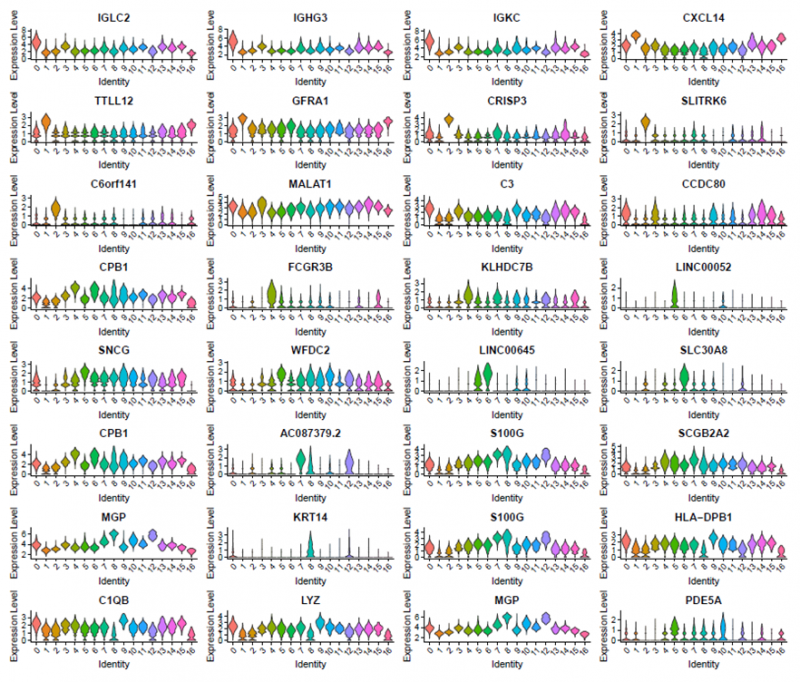
图 14 所有 cluster 选择 top5 基因绘制小提琴

图 15 特定 pathway 功能富集分析
10X Genomics Visium 应用方向
1、应用方向
空间位置信息,或者细胞在组织中天然的状态在研究过程中其实具有十分重要的价值,特别针对某些研究领域,如发育生物学(不同位置的细胞接受不同的信号浓度梯度、响应不同的外界刺激,具有不同的发育命运)、肿瘤生物学(肿瘤组织与癌旁组织的区别,肿瘤细胞侵润过程中肿瘤细胞的变化与对正常细胞的影响,肿瘤转移的不同过程阶段等)、脑神经科学(不同脑区位置的神经元结构、神经连结,中间神经元投射,突触前后,神经胶质相互影响等等),细胞来源的位置信息是极为关键的决定因素。常见空间转录组的应用方向主要在肿瘤学,免疫学,发育生物学,神经科学及病理学等方向。

图 16 空间转录组的应用方向
表 1 10X Genomics 空间转录组已发表文献
|
编号 |
期刊 |
年份 |
物种 |
研究方向 |
题目 |
|
1 |
Cell |
2019 |
人 |
胚胎心脏 |
A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart[4] |
|
2 |
Science |
2019 |
人、小鼠 |
肌萎缩性侧索硬化症 |
Spatiotemporal dynamics of molecular pathology in amyotrophic lateral sclerosis[5] |
|
3 |
Scientific Reports |
2019 |
人 |
关节炎 |
Exploring inflammatory signatures in arthritic joint biopsies with Spatial Transcriptomics[6] |
|
4 |
Bioinformatics |
2019 |
|
方法 - 分析和可视化工具 |
ST viewer: a tool for analysis and visualization of spatial transcriptomics datasets[7] |
|
5 |
Nature Communications |
2018 |
人 |
前列腺癌 |
Spatial maps of prostate cancer transcriptomes reveal an unexplored landscape of heterogeneity[8] |
|
6 |
Bioinformatics |
2018 |
|
方法 -ST-Spot 检测器 |
ST Spot Detector: a web-based application for automatic spot and tissue detection for spatial Transcriptomics image datasets[9] |
|
7 |
Scientific Reports |
2018 |
人 |
牙周炎 |
Gene expression profiling of periodontitis-affected gingival tissue by spatial transcriptomics[10] |
|
8 |
Cancer Research |
2018 |
人 |
皮肤恶性黑色素瘤 |
Spatially Resolved Transcriptomics Enables Dissection of Genetic Heterogeneity in Stage III Cutaneous Malignant Melanoma[11] |
|
9 |
Nature Protocols |
2018 |
|
方法 - 条码固相 RNA 捕获 |
Barcoded solid-phase RNA capture for Spatial Transcriptomics profiling in mammalian tissue sections[12] |
|
10 |
Nature Plants |
2017 |
拟南芥、杨树云杉 |
花序、叶芽和雌球果发育 |
Spatially resolved transcriptome profiling in model plant species[13] |
|
11 |
Scientific Reports |
2017 |
人 |
心脏 |
Spatial detection of fetal marker genes expressed at low level in adult human heart tissue[14] |
|
12 |
Science |
2016 |
|
方法 - 芯片技术 |
Visualization and analysis of gene expression in tissue sections by spatial transcriptomics[15] |
|
13 |
Nature Communications |
2016 |
|
方法 -MASC-seq |
Massive and parallel expression profiling using microarrayed single-cell sequencing[16] |
|
14 |
Scientific Reports |
2016 |
|
方法 - 文库制备 |
An automated approach to prepare tissue-derived spatially barcoded RNA-sequencing libraries[17] |
表 2 10X 官方已做过优化的样本类型
|
人组织类型 |
小鼠组织类型 |
大鼠组织类型 |
||||
|
组织类型 |
疾病状态 |
切片厚度 |
组织类型 |
切片厚度 |
组织类型 |
切片厚度 |
|
大脑 |
恶性胶质瘤 |
10-25µm |
眼睛 |
10µm |
大脑 |
10-25µm |
|
乳腺 |
健康 |
10µm |
大肠 |
10µm |
心脏 |
10µm |
|
乳腺 |
浸润性导管癌 |
10µm |
肝脏 |
10µm |
肾脏 |
10µm |
|
乳腺 |
浸润性小叶癌 |
10µm |
肺 |
10µm |
伯豪生物测试 样本类型 |
|
|
心脏 |
健康 |
10µm |
嗅球 |
10µm |
||
|
肾脏 |
健康 |
10µm |
卵巢 |
10µm |
大脑 |
10µm |
|
肾脏 |
肾炎 |
10µm |
四头肌 |
10µm |
小脑 |
10µm |
|
大肠 |
结肠癌 |
10µm |
小肠 |
10µm |
心脏 |
10µm |
|
肺 |
乳头状癌 |
10µm |
脾脏 |
10µm |
肝脏 |
10µm |
|
淋巴结 |
健康 |
10µm |
胃 |
10µm |
脾脏 |
10µm |
|
淋巴结 |
发炎 |
10µm |
睾丸 |
10µm |
肾脏 |
10µm |
|
脾脏 |
发炎 |
10µm |
甲状腺 |
10µm |
肺 |
10µm |
|
卵巢 |
肿瘤 |
10µm |
舌 |
10µm |
…… |
…… |
2、案例解析
案例一
基于空间转录组测序进行肌萎缩性侧索硬化症分子病理学的时空动力学研究
Spatiotemporal dynamics of molecular pathology in amyotrophic lateral sclerosis[5]
发表杂志:Science
影响因子:41.037
发表单位: 美国纽约基因组中心等
发表时间:2019 年 4 月 5 日
摘要:
肌萎缩性脊髓侧索硬化症(ALS) 的瘫痪是由运动神经元退化导致的骨骼肌失神经引起的。运动神经元和神经胶质之间的相互作用导致了运动神经元的丢失,但是在完整的脊髓组织中驱动这些过程的分子事件的时空顺序仍然知之甚少。在这里,作者使用空间转录组学来获得疾病过程中小鼠脊髓的基因表达量,以及肌萎缩性脊髓侧索硬化症患者死后组织的基因表达量,以描述肌萎缩性脊髓侧索硬化症的潜在分子机制。通过识别通路动力学,在早期区分小胶质细胞和星形胶质细胞种群的区域差异,并识别小鼠 ALS 模型和人类死后脊髓之间共享的几个转录通路的扰动。
研究方法:
作者采用 ALS 小鼠(G93A)和野生型小鼠(WT),取发病前、早期症状开始、有症状和疾病末期等四个时间点的腰椎脊髓组织(L3—L5)进行空间转录组测序,获得基因表达的空间转录图谱,然后采用 ALS 患者死后的腰椎和颈椎脊髓的组织切片进行空间转录组测序来描绘基因的表达。作者收集了来自 67 只小鼠 1165 个组织切片的 76136 个空间基因表达测量值(spatial gene expression measurement,SGEM)和来自 7 名 ALS 患者 80 个组织切片的 61031 个 SGEM。作者使用解剖注释区域(anatomical annotation region,AAR)tags 对每一个 SGRM 进行注释,并利用这些 tags 进行差异基因表达分析。为了准确评估条件内外基因的表达水平、检测显著性区域以及解剖学和细胞类型上的改变,作者开发出分层生成概率模型(hierarchical generative probabilistic model),基于空间单细胞转录组数据学习,对小鼠脊髓的 11,138 个基因和人类脊髓的 9624 个基因的空间分布进行可靠的定量 PCA 分析 SGEM 数据的变化主要受到空间位置、疾病状态和基因型的影响。作者针对小鼠空间转录组数据开发了无偏共表达分析(unbiased coexpression analysis),来鉴定主要的共表达基因模块,分别代表不同的时空表达和通路活动。利用以上新的计算方法,作者揭示了疾病驱动的中枢神经系统中所有细胞类型的多个信号通路的活性变化。
研究内容:
小鼠脊髓基因的时空表达;ALS 发生早期 TREM2- 和 TYROBP- 介导的信号通路异常;小鼠 ALS 发展过程中基因表达的时空动态变化;人类 ALS 患者脊髓组织时空转录组分析。

图 17 小鼠脊髓基因的时空表达
(A 上)小鼠腰椎解剖区域(AARs)HE 染色图;(A 左)基于 WT 小鼠 SGEMs 的 mRNA 区域空间表达可视化图;(A 右)共聚焦免疫荧光图像;(B)产后 70 天(P70)和 100 天(P100)小鼠 Aif1 和 Gfap 基因的空间 mRNA 表达情况;(C)P70 和 P100 小鼠 AIF1 和 GFAP 蛋白共聚焦免疫荧光图像
案例二
空间分辨率的转录组学能够解析 III 期皮肤恶性黑色素瘤的遗传异质性
Spatially Resolved Transcriptomics Enables Dissection of Genetic Heterogeneity in Stage III Cutaneous Malignant Melanoma[11]
发表杂志:Cancer Research
影响因子:8.556
发表单位: 瑞典皇家理工学院基因技术系;瑞典卡罗林斯卡医学院肿瘤科 - 病理学系;发表时间:2018 年 10 月 15 日
摘要:
皮肤恶性黑色素瘤(黑色素瘤)的特征是高突变负荷,广泛的肿瘤间和瘤内遗传异质性,以及复杂的肿瘤微环境(TME) 相互作用。深入了解黑色素瘤的机制对于理解肿瘤的进展和治疗反应是至关重要的。作者将空间转录组学(ST) 技术应用于黑色素瘤淋巴结活检,并成功地对 2200 多个组织域的转录组进行了测序。反褶积技术与传统的转录组数据降维方法相结合,既能可视化组织内的转录景观,又能识别与特定组织学实体相关的基因表达谱。无监督分析显示了一个复杂的空间瘤内组成的黑色素瘤转移,这是不明显的形态注释。每个活检都显示了不同的基因表达谱,包括多个黑色素瘤特征在一个肿瘤区域内共存的例子,以及根据其空间位置和基因表达谱特征的淋巴组织共享的基因表达谱。接近肿瘤区域的淋巴样区表现出一种特殊的表达模式,这可能反映了 TME,这是全面了解肿瘤进展的关键组成部分。总之,使用 ST 技术来生成基因表达谱可以揭示黑色素瘤转移的详细情况。这将激励研究人员将空间信息整合到分析中,以识别肿瘤进展和治疗结果的潜在因素。
研究方法:
该研究包括来自 4 名 IIIc 期黑素瘤患者的淋巴结转移的样本。根据 Johansson 及其同事的结果分类(>60 个月),除一个样本外,其他所有样本均为 BRAFv600 阳性,一名患者为长期幸存者。ST 分析前的 RNA 质量控制显示 RNA 完整性数(RIN)在 6.2 和 7.4 之间。由训练有素的病理学家进行的手动组织学注释鉴定了转移灶内肿瘤细胞(即黑素瘤),基质和淋巴样的区域。注释结果用于与 ST 基因表达分析的结果进行比较。
该研究将空间转录组学(ST)技术应用于黑素瘤淋巴结活体检测,并成功测序了超过 2,200 个组织结构域的转录组。解卷积(Coevolution)与传统的转录组数据降维算法相结合,使作者既可以观察组织内的转录图谱,又可以识别与特定组织学实体相关的基因表达谱。作者的无监督分析(Unsupervised Analysis)通过对形态学注释不明显的每个活体单元检测显示出不同的基因表达谱,证明黑色素瘤转移过程中具有复杂空间肿瘤内成分,包括单个肿瘤区域内多个黑素瘤特征共存的实例以及根据其空间位置和基因表达谱表征的淋巴组织的共享特征。
研究内容:
淋巴结转移的空间分析步骤;从黑素瘤淋巴结转移产生空间分辨的转录组数据;通过因子分析和 t -SNE 可视化 2200 个组织结构域中的基因表达;肿瘤微环境 TME 中基因表达的可视化。

图 18 空间转录组(ST)方法的概述
空间转录组的过程总览以及下游的分析,带有条形码的微阵列包含 1007 个提前包被带有独特的空间 ID 条形码(Unique Barcode)RT 引物的点格,每个点格的直径在 100µm,这个可以和组织的空间结构域相对应,区域中心和区域中心的距离为 200µm。
案例三
人心脏发育过程中全器官的基因表达和细胞图谱
A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart[4]
发表期刊:Cell
影响因子:36.216
发表单位: 瑞典皇家理工学院基因技术系;冷泉港实验室等
发表时间:2019.12.12
摘要:
人类心脏形态发生的过程尚不完全清楚。它的完整特征需要通过深入探索器官范围的单细胞空间分辨率的基因表达谱。在此,作者提出了一种分子方法,它揭示了胚胎心脏在三个发育阶段的细胞类型的全面转录图景,并将特定细胞类型的基因表达映射到特定的解剖结构域。通过单细胞空间转录组测序鉴定人胚胎心肌细胞类型,证实并丰富了胚胎心脏基因表达的空间注释。然后使用原位测序来提炼这些结果,并创建三个发育阶段的空间亚细胞图。最后,作者创建了一个公开可用的人类发育心脏的网络资源,以促进未来对人类心脏发生的研究。
研究方法:
分别在 4.5~5、6.5、6.5~7 和 9 PCW 这 4 个时间点采集 4 种发育心脏组织,受孕后年龄和临床年龄通过临床超声和胚胎的分期解剖学标志确定。计算得出心脏组织的性别信息。具体地说,4.5- 5 和 6.5 PCW 的心脏是男性,而 6.5- 7 和 9 PCW 的心脏是女性。对 4.5、6.5 和 9PCW 的心脏进行 ST 和 ISS 分析,而 6.5-7PCW 的心脏进行 scRNAseq。将 6.5 和 6.5-7 PCW 的样本作为生物重复样本,比较 ST 和 scRNA-seq 检测到的基因表达。在 ST 和 ISS 实验中,来自同一心脏组织的连续组织切片被认为是技术上的重复。但是,重要的是要注意,连续的部分高度相似,但不完全相同。共计 2 个 scRNA-seq 和 19 个 ST。
研究内容:
人心脏发育过程中基因表达的时空动力学;单细胞转录组测序研究心脏发育不同阶段异质性;构建发育中的心脏表达图谱;通过时空分析解决细胞类型相似性问题。
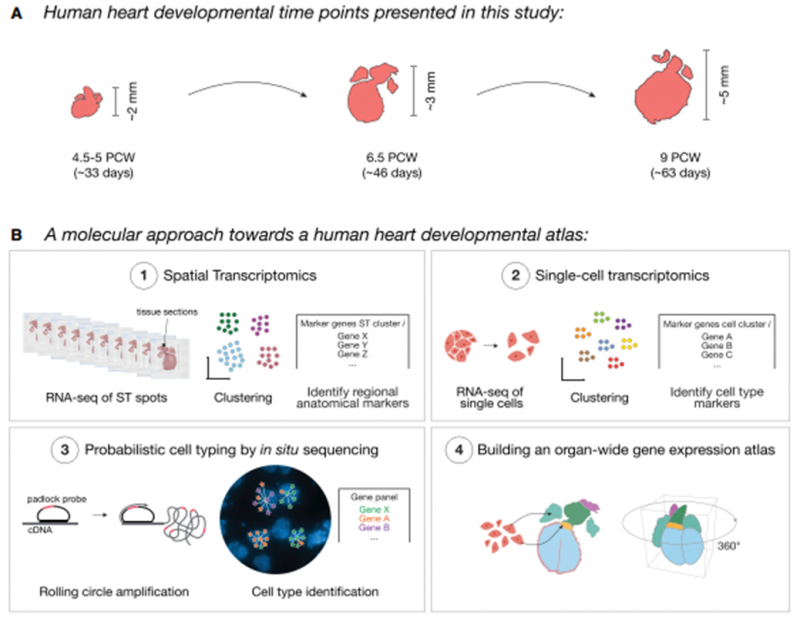
图 19 研究概况
A、本研究包括三种人类心脏组织的发育阶段。B、本研究所采用的分子生物学方法:(1)利用空间转录组技术(ST),鉴定出了解剖学区域特异的 marker;(2)用 scRNA-seq 技术分析心脏中间时间点的细胞类型异质性;(3)用 ISS 技术定位亚细胞分辨率的关键基因;(4)构建器官水平的 3D 基因表达图谱。
样品准备
1、组织冷冻
(1)异戊烷和液氮浴:如图所示,不锈钢烧杯内倒入异戊烷至 2 / 3 体积,然后将不锈钢烧杯放入液氮中(与异戊烷液位相同),孵育 15 分钟。
(2)新鲜样本可用 PBS 冲洗,去除残留血液,然后使用实验室纸巾吸干组织表面多余的血液或液体,防止冰晶的形成。
注:组织长和宽不可超过 6mm±0.2mm,否则后续实验无法贴片。
(3)用镊子或刮刀将组织完全浸没在异戊烷中,直至完全冰冻。
注:冷冻时间可根据组织类型和大小而改变。
(4)冷冻后,取出组织转移到预冷的密封容器中,干冰转移至 -80°长期保存或立即进行下一步。
注:为防止组织样品蒸发和脱水,冷冻的组织样品必须储存在密封容器中以长期保存。
2、冷冻组织包埋
(1)粉状干冰:用研钵和杵准备干冰粉。
(2)冷冻 OCT:将 OCT 放在冰上≥30 分钟。
注:OCT 请使用指定品牌
(3)预冷镊子:将镊子放在干冰中≥30 分钟。
(4)包埋盒上需标记组织样本的方向。
注:在添加 OCT 和组织之前,需先在包埋盒上标记,因为一旦冻结,OCT 将迅速变成白色,这使得以后很难确定组织方向。
(5)用冷却的 OCT 铺平包埋盒底部,避免产生气泡。
(6)从不锈钢烧杯中取出冰冻的组织,加入到 OCT 的包埋盒中心位置,继续倒入 OCT 完全覆盖样本。需避免气泡产生,尤其在组织附近。
(7)立即将含有组织和 OCT 的包埋盒放在干冰粉上,直至完全冻结(约 20min 以上)。
(8)将包埋盒放置到密封袋中,干冰运输。


